Computational approach to distinguish functional protein patterns between pediatric and adult Actute Myeloid Leukemia
Fieke W. Hoff
UMC Groningen – Kinderoncologie/Hematologie
In januari 2016 ben ik voor 7 maanden naar het precieuze MD Anderson Cancer Center in Houston (USA) geweest om onderzoek te doen naar eiwit expressie profielen in kinderen met acute leukemie. Acute leukemie is de meest voorkomende vorm van kanker bij kinderen en blijft ondanks grote vooruitgang over de laatste tientallen jaren in behandelstrategieën een levensbedreigende ziekte met een overlevingspercentage van ~90% voor acute lymfoblastische leukemie (ALL) en slechts 60-70% voor acute myeloïde leukemie (AML). Heterogeniteit in het genetische landschap van acute leukemie maakt bijna elke bijna patiënt genetisch uniek, wat het vinden van geïndividualiseerde behandelingen complex maakt. Omdat genetica tot uiting komt via de expressie en activatie van eiwitten, veronderstelden wij dat deze genetische heterogeniteit samengaat met een bepaald aantal eiwit expressie signaturen, die uiteindelijk kunnen leiden tot meer toegespitste therapie.
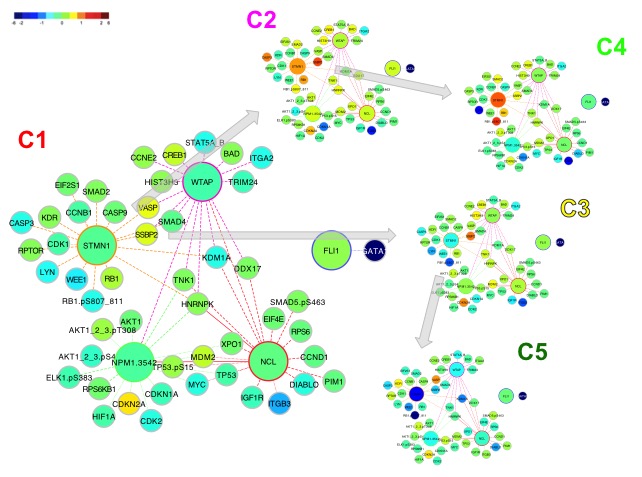
Relatieve eiwit patronen werden bepaald met behulp van Reverse Phase Protein Arrays (RPPA) uitgevoerd met samples van kinder-ALL en -AML en 10 normale CD34+ beenmerg samples; deze techniek maakte mogelijk om gelijktijdig op grote schaal de eiwit expressie te bepalen met behulp van 194 antilichamen gericht tegen het gehele eiwit of specifieke activatie domeinen. Omdat eiwitten in de cel functioneren in een netwerk waarbij er interactie plaatsvindt met andere eiwitten, zijn de eiwitten eerste ingedeeld op basis van hun functie of interacties in de cel zoals bekend in de literatuur. Dit leidde tot de vorming van 31 Eiwit Functionele Groepen. Patiënten werden hierna geclusterd per functionele eiwit groep op basis van hun expressie, wat leidde tot de vorming van een optimaal aantal Eiwit Clusters per eiwit functionele groep; bepaalde eiwit clusters lieten verhoogde eiwit expressie zien ten opzichte van normaal beenmerg, terwijl andere eiwit clusters een verminderde expressie aantoonden. Vervolgens hebben we netwerken gemaakt voor elk eiwit cluster met behulp van bekend interacties uit de literatuur en sterke associaties in de literatuur (Figuur). Deze netwerken toonden verschillende overgangsstaten aan en onthulden “normaal-gelijkende” en “leukemie-specifieke” eiwit patronen. Hiërarchische clustering van een compilatie van alle eiwit clusters stelde sterk co-optreden tussen verschillende eiwit clusters vast en toonde het bestaan aan van eiwit constellaties; een groep van eiwit clusters van verschillende eiwit functionele groepen die sterk met elkaar correleren. Patiënten met hetzelfde patroon van eiwit constellaties vormden samen een eiwit expressie signatuur.
Met het idee dat deze signaturen patiënten kunnen classificeren en kunnen leiden tot meer gerichtere en geïndividualiseerde therapie, is daarna voor elke constellatie en elk signatuur bepaald welke eiwitten een verhoogde expressie hadden en welke een verminderde expressie. Deze eiwitten hebben we geselecteerd en verdere experimenten moeten nu aantonen of combinaties van deze cruciale eiwitten in combinatie met bestaande therapieën gevalideerd kunnen worden als target voor specifieke eiwit expressie signaturen. Omdat we deze methode zowel toegepast hebben in ALL en AML bij kinderen, biedt dit de kans om deze twee aandoeningen met elkaar te vergelijken. Dit zal inzicht geven in welke eiwit profielen specifiek zijn voor ALL of AML en welke voorkomen in beide ziekten. Daarnaast biedt een eerdere studie de kans om eiwit profielen van AML te vergelijken met die in volwassenen.
Dankzij de goede samenwerkingen tussen de het lab van de kinderoncologie onder leiding van Prof. de Bont in het UMCG en het lab van Prof. Kornblau in het MDACC, heb ik deze ultieme kans gekregen om aanraking te komen met wetenschappelijk onderzoek in een inspirerend en vooraanstaand ziekenhuis. Daarnaast heeft deze stage inzicht gegeven in wat er allemaal speelt binnen het onderzoek naar leukemie. Kortom, een ontzettend interessante en leerzame ervaring, die mij stimuleert verder te gaan met onderzoek naar acute leukemie!